Die umfassende molekulare Vielfalt entsteht aber erst auf der Ebene der Proteine, deren Gesamtheit aller Proteine in einem Lebewesen, einem Gewebe, einer Zelle oder einem Zellkompartiment unter exakt definierten Bedingungen und zu einem bestimmten Zeitpunkt als Proteom bezeichnet wird. Eine Modellrechnung ergab: Wenn man von einer durchschnittlichen Peptidlänge eines Eiweißes von 200 Aminosäuren ausgeht, könnten bei einem Vorrat von 20 Aminosäuren 10260verschiedene Eiweiße synthetisiert werden.
Nun könnte man erwarten, daß die Kenntnis der Transkriptionsmuster mit der Kenntnis der zu erwartenden Proteinmuster gleichzusetzen ist. Dies ist offensichtlich nicht der Fall. Zwischen Transkriptom und Proteom bestehen große Unterschiede. Es gibt nur schwache Korrelationen zwischen Boten-RNS- und Protein-Expressionsmustern. So korrelieren die dynamischen Veränderungen auf der Ebene der mRNS-Transkription nur zu einem gewissen Prozentsatz mit der Zahl der resultierenden Proteine. Man weiß heute, daß bereits in einer frühen Phase des Weges vom Gen zum Eiweiß allein durch das Zusammenwirken verschiedener Gene und das unterschiedliche Zusammensetzen der Exons nach der Entfernung der Introns (Spleißen) die Voraussetzung für die Synthese unterschiedlicher Eiweiße geschaffen wird. Ohne diese Prozesse wäre z.B. die Synthese hochspezifischer Antikörper nach einer Infektion gar nicht denkbar (vgl. Abschnitt „Vom Gen zum Eiweiß“).
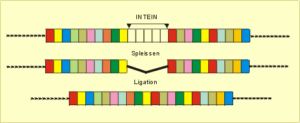
Noch aufregender ist das erst seit kurzer Zeit bekannte Spleißen auf Proteinebene (!), dessen Prinzip in Abb. 18 dargestellt ist. Es gibt Proteine, die über eine spezielle Aminosäuresequenz (sog. Inteine) innerhalb der langen Peptidkette verfügen, die sich selbst enzymatisch ausschneiden und dabei die entstehenden Peptidenden miteinander verbinden können (Ligation). Auf diese Weise werden die entstandenen Proteinfragmente zu neuen Proteinkombinationen zusammengefügt. Dieses Verfahren läßt sich auch ausserhalb der lebenden Zelle im Reagenzglas anwenden und ermöglicht so die Herstellung „künstlicher“ Eiweißvarianten mit unterschiedlicher Funktionalität wie jüngst in der Zeitschrift „Nature“ beschrieben wurde. Doch damit nicht genug. Unter bestimmten Bedingungen sind verschiedene eiweißspaltende Enzyme in der Lage, in umgekehrter Richtung zu wirken (Reverse Proteolyse) und Proteinfragmente nicht etwa zu spalten, sondern zusammenzufügen, ein Phänomen, das hauptsächlich bei Pflanzen beobachtet wurde.
Auf diese und andere Weise erhöht sich also die Zahl der theoretisch möglichen Eiweiße um ein Vielfaches. Man rechnet auf ein Gen mindestens zehn verschiedene Eiweiß-Endprodukte. Dies bedeutet für die rund 25.000 kodierenden Gene eine theoretische Gesamtzahl von mehr als 250.000 verschiedenen Proteinen.
Erinnern wir uns an die Ausführungen über die die Struktur der Eiweiße, wo wir erfahren haben, daß es sich bei den Eiweißen um hochdynamische dreidimensionale Strukturen handelt, deren räumliche Konfiguration stark vom Milieu abhängt, das sie zu einem bestimmten Zeitpunkt umgibt. Das Milieu bestimmt dabei die Ladungsmuster an der Oberfläche der einzelnen Moleküle und diese wiederum die Wechselwirkungsmöglichkeiten mit anderen Molekülen. Ihre Kenntnis ist von allergrößter Bedeutung. Hier werden hauptsächlich die Ansatzpunkte gesehen, um Krankheitsursachen zu erkennen und diese pharmakologisch zu beeinflussen.
Als Fortsetzung des internationalen Genomprojekts wurde ein weiteres Konsortium , die Human Genom Proteom Organisation, HUPO ) gegründet, die es sich zur Aufgabe gemacht hat, bis zum Jahre 2007 alle Proteine des Menschen und seine Wechselwirkungsmöglichkeiten zu bestimmen . Die Deutsche Gesellschaft für Proteomforschung e.V. (DGPF) wurde 2001 in Martinsried im Max Planck Institut für Biochemie gegründet.
Der enorme Rechenaufwand eines solchen Proteomprojekts, der für die Struktur- und Wechselwirkungsanalyse erforderlich ist, hat zwangsläufig zu neuen Kooperationen zwischen Gentechnik-Firmen und großen Computerkonzernen geführt, die mit vernetzten Riesencomputern den gewaltigen Aufgaben stellen wollen. Aufgrund der nicht vorhersehbaren Ausmaße der Untersuchungen haben sich die strategischen Ziele heute jedoch auf ein eher realistisches Maß beschränkt. So wird man sich in der nächsten Zukunft tendenziell auf wenige pathologisch und pharmakologisch wichtige Proteine und deren molekulare Wechselwirkungen beschränken (müssen).
Fazit: Die Zahl aller synthetisierten Proteine (das Proteom) ist nicht identisch mit den primär abgelesenen genetischen Informationen, dem Transkriptom. Durch das Zusammenwirken verschiedener Gene und das unterschiedliche Zusammensetzen der Exons nach der Entfernung der Introns (Spleißen) sowie der zahlreichen epigenetischen Einflüsse wird die Voraussetzung für die Synthese vieler zusätzlicher und unterschiedlicher Eiweiße geschaffen. Die Kenntnis der Syntheseprodukte und deren pharmakologische Wechselwirkungsmöglichkeiten bilden die Grundlage für die Entwicklung neuer Pharmaka und Behandlungsmöglichkeiten verschiedener Erkrankungen.
