Fast jedermann weiß heutzutage aus dem Schulunterricht, dass der Hauptteil der Erbinformationen im Kern der Zelle in den Chromosomen lokalisiert ist und über die Keimzellen (Spermien, Eizellen) an die Nachfahren weitergegeben wird. Die Träger der Erbinformationen – die Nukleinsäuren, namentlich die DNS – sind in aller Munde (genetischer Fingerabdruck, Präimplantationsdianostik, Klonen u.a.).
Im Folgenden werden wir uns kurz mit den Nukleinsäuren, deren Aufbau und Funktion sowie dem Zusammenhang zwischen den Erbinformationen und der Biosynthese von Eiweißen beschäftigen, um herauszufinden, wie und wo die Informationen für die individuellen Unterschiede von Mensch zu Mensch gespeichert sind. Doch zuerst ein wenig Basiswissen.
Bausteine. Die molekulare Grundlage der Nukleinsäuren (Kernsäuren) bilden sehr kleine organische Bausteine sog. Nukleotide, die sich jeweils aus drei Molekülen zusammensetzen: einem basischen Ringmolekül (Base), einem Zucker- und einem Phosphatrest. Entsprechend der Art des Zuckermoleküls (Ribose oder Desoxyribose) werden Ribonukleinsäuren (RNS; engl.: RNA – ribonucleic acid) und Desoxyribonukleinsäuren (DNS; engl.: DNA – desoxyribonucleic acid) unterschieden. Im vorliegenden Text verwenden wir die deutsche Schreibweise (DNS und RNS).
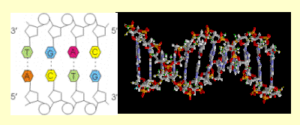
Abb.6 zeigt in einem vereinfachten zweidimensionalen Schema die Anordnung der einzelnen Nukleotide in einem Nukleinsäuremolekül und deren resultierende dreidimensionale Helixstruktur. Die für einen Nichtchemiker vielleicht abschreckende Strukturformel ist für das weitere Verständnis nicht erforderlich. Das Wissen um die Bedeutung der Nukleotide, die sozusagen die Buchstaben des genetischen Kodes darstellen, sollte der Leser jedoch für das Verständnis der nächsten Kapitel ständig zur Verfügung haben.
Es gibt vier verschiedene Basen, die vier verschiedene Nukleotid-Bausteine der DNS charakterisieren: Adenin (A), Guanin (G), Thymidin (T) und Cytosin (C). Im Falle der RNS steht Uridin (U) anstelle von Thymidin (T). Die sequenzielle Abfolge dieser vier Nukleotide bildet die Grundlage für den genetischen Kode, die Basis der molekularen Genetik. Dieses Programmiersystem funktioniert universell in der belebten Natur – vom Grippevirus bis zum Menschen!
Struktur. Lange Molekülketten aus Millionen oder Milliarden von Nukleotiden, die über den Zucker- bzw. Phosphatanteil miteinander verknüpft sind, bilden die Primärstruktur der Nukleinsäuren (Abb. 7 links). Neben der bloßen Aneinanderreihung der Nukleotide weisen die Nukleinsäuren eine strenge räumliche Spiralstruktur – die Sekundärstruktur – auf, die aus zwei Einzelsträngen besteht, in der sich jeweils zwei komplementäre Basen gegenüberstehen (komplementäre Basenpaarung), nämlich G gegenüber C und T gegenüber A. Der erst vor wenigen Jahren verstorbene Erwin Chargaff hatte bereits 1950 herausgefunden, dass in jeder DNS die Bausteine A und T bzw. G und C jeweils im exakt gleichen Mengenverhältnis vorkommen. So lag die Vermutung nahe, dass es sich bei den Nukleinsäuren um eine komplementäre Molekülstruktur handeln musste.
Die beiden Einzelstränge werden über schwache Wasserstoffbrücken (nicht über feste chemische Bindungen) zwischen den komplementären Basen zusammengehalten. Auf diese Weise entsteht ein gegenläufiger Doppelstrang (Doppelhelix, Abb. 7 rechts), der entweder mit einem Zucker- oder Phosphatrest endet. Die Stabilität des Riesenmoleküls wird durch das Milieu der Zelle erreicht, das dem des Urozean ähnelt, aus dem alles Leben einstmals hervorgegangen ist.
Am 25. April 1953 veröffentlichten die Wissenschaftler James Watson und Francis Crick anhand eines räumlichen Modells ihre Hypothese von der DNS-Doppelhelix. Für dieses Modell erhielten sie 1962 den Nobelpreis für Medizin und Physiologie. Titel: „Entdeckung über die Molekularstruktur der Nukleinsäuren und ihre Bedeutung für die Informationsübertragung innelebender Substanz“.
Um sich eine Vorstellung von den Dimensionen des universellen Informationsträgers DNS machen zu können, hier noch einige erstaunliche Fakten: Die Länge der DNS in den 46 Chromosomen eines einzelnen menschlichen Zellkerns beträgt in der Summe etwa 2 Meter. So ist der DNS-Faden eine Milliarde mal länger als sein Durchmesser. Zieht man alle Zellen des Körpers in Betracht, so kommt man auf eine Gesamtlänge des DNS-Fadens von 200 Milliarden Kilometer, also der siebzigfachen Strecke Sonne-Saturn. Allein die Tatsache, dass die 2 m DNS in einer einzelnen Zelle im winzigen Zellkern untergebracht werden muss und zum Zeitpunkt der Zellteilung wieder aufgerollt wird, um danach erneut in die kompakte Form gebracht zu werden, sollte uns ein wenig Ehrfurcht vor dem Leben einflößen.
Vervielfältigung (Amplifikation). DNS aus einem biologischen Material zu isolieren ist kinderleicht und bedarf keines größeren labortechnischen Aufwandes. In vielen Fällen, insbesondere bei der Bearbeitung kriminalistischer Fragestellungen mit geringsten Materialspuren, ist es jedoch erforderlich, die zur Verfügung stehende Probenmenge zu vervielfältigen, um in den nachfolgenden Nachweisverfahren zuverlässige Resultate zu erzielen.
Das Prinzip der DNS-Vervielfältigung mit Hilfe der Polymerase-Kettenreaktion (PCR) ist leicht zu verstehen: Die Doppelstränge der DNS werden durch Hitze (ca. 95°) aufgespalten. Durch Zugabe von Nukleotidbausteinen (A,T,G,C) und eines Enzyms (Polymerase) werden die komplementären Gegenstränge der beiden Einzelstränge bei niedrigeren Temperaturen synthetisiert.
Die Methode wurde bereits Mitte 1985 von Karry Mullis entwickelt, für die er 1993 den Nobelpreis für Chemie erhielt. Sie ahmt praktisch die identische Verdopplung der DNS in kernhaltigen Zellen bei der Zellteilung nach. Mittels PCR kann durch etwa 30 solcher Zyklen die DNS-Vorlage aus der Probe auf das Milliardenfache vermehrt werden. Die PCR kann eingesetzt werden, um mit Hilfe sogenannter Primer (Startsequenzen) einen kurzen, genau definierten Teil eines DNS-Strangs zu vervielfältigen. Dabei kann es sich um ein Gen oder auch nur um einen Teil eines Gens oder einen nichtkodierenden Abschnitts der DNS (wie beim genetischen Fingerabdruck) handeln. Im Gegensatz zu lebenden Organismen kann der PCR-Prozess nur kurze DNS-Abschnitte bis zu 10.000 Basenpaaren kopieren.
Eine Zufallsentdeckung hat die weite Verbreitung dieses Verfahren möglich gemacht. Wie gesagt, benötigt man zur Amplifikation ein Enzym, das bei einer Temperatur von 95°C stabil und wirksam ist und nicht wie die meisten Eiweiße bei 60°C seine Arbeit einstellt. So entdeckte man ein Bakterium namens Thermophilus aquaticus, das bei über 110°C in Geysiren des Yellowstone National Parks (USA) lebt und dessen Enzyme außerordentlich hitzestabil sind. Der Name der heute meist benutzten Polymerase leitet sich deshalb von dem Namen dieses Bakteriums ab: Taq-Polymerase.
Fazit: Die Nukleinsäuren DNS und RNS sind aus vier verschiedenen Nukleotidbausteinen aufgebaut. Ihre Aufeinanderfolge bildet den universellen genetischen Kode für die Synthese der verschiedenen Eiweiße. Die Nukleinsäuren weisen eine strenge räumliche Spiralstruktur (Doppelhelix) auf, die aus zwei Einzelsträngen besteht.
